10/11/2009
Mutações e o escape da imunidade
Se sarampo é causado por um vírus e só se pega uma vez na vida, por que podemos ficar gripados todos os anos?
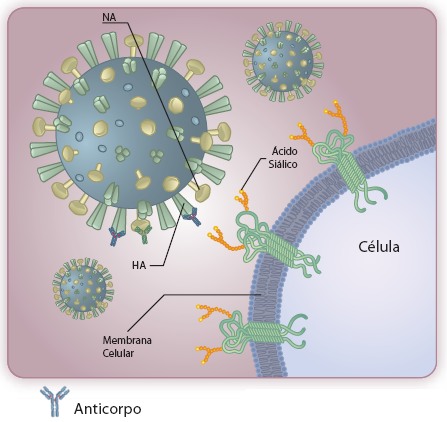
Anticorpo ligado à Hemaglutinina (HA) a impede de se ligar ao ácido siálico da célula.
Quando ficamos gripados, em poucos dias o corpo parece se livrar do vírus. Raramente os sintomas chegam a durar duas semanas e, um período maior costuma indicar complicações causadas por outros microorganismos. Este período curto da gripe se dá por conta de nossa resposta imune.
Conforme o vírus se replica no corpo de pessoas infectadas, o sistema imune captura vários pedaços de proteínas virais, os chamados antígenos, e produz anticorpos contra elas. Há também outros tipos de reposta imune, mas que não causam imunidade de longo prazo, de maneira que não vêm ao caso. Os dois maiores alvos virais dos anticorpos são a Hemaglutinina e a Neuraminidase, uma vez que são as proteínas mais expostas do vírus. Quando os anticorpos se ligam a elas, além de sinalizarem para macrófagos e outros tipos de células de defesa que aquele corpo estranho (o vírus) tem de ser atacado, ainda podem impedir o funcionamento do vírus. Um anticorpo que ataque região de reconhecimento do receptor da Hemaglutinina impede que ela se ligue às células. São os chamados anticorpos neutralizantes.
Graças a esta resposta imune, em alguns dias aquele Influenza não consegue mais nos infectar. Mas como o vírus consegue voltar?
Como já vimos antes, ao replicar o seu genoma, a polimerase do Influenza provoca uma série de mutações. Estas mutações mudam a composição das proteínas virais. Quando são trocados os aminoácidos – componentes das proteínas – da região em que o anticorpo se liga, ou antígeno, ele pode perder a afinidade. Assim, conforme o vírus infecta novos hospedeiros vai acumulando pequenas mudanças que ao final terão mudado seus antígenos. Este processo é chamado de variação antigênica gradual ou antigenic drift.
Muitas vezes estas mudanças não precisam ser bruscas para fazer efeito. Mudanças em locais importantes para o reconhecimento pelo anticorpo de apenas um aminoácido podem ser suficientes. Isso foi proposto por um dos trabalhos mais importantes sobre a imunologia do Influenza já feitos, que mudou a forma como estudamos o vírus.
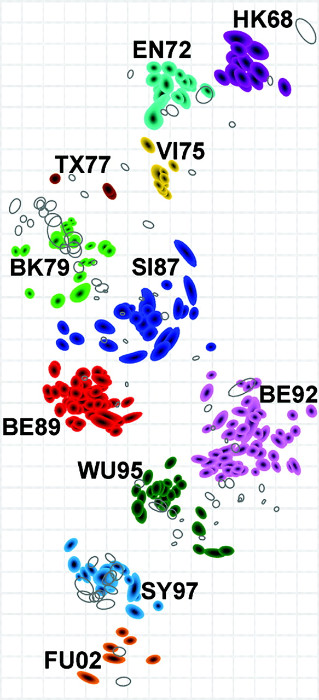
Mapa antigênico da Hemaglutinina do H3N2 coletado de 1968 (ano de entrada dele em humanos) a 2003. A cor dos círculos define o grupo a que aquela amostra está relacionada. Cada unidade de distância entre eles representa a diminuição da resposta imune ao vírus em relação aos anticorpos produzidos contra o anterior. Assim, quanto mais distante um vírus A estiver do B, menor a reconhecimento dele pelos anticorpos contra B.
Utilizando uma idéia proposta em 2001, ao invés de olharem apenas para a diferença genética entre amostras do H3N2 causador da gripe humana de anos diferentes, os autores resolveram levar em conta a resposta imune contra eles. Com isso, são construídos mapas que apontam quão bem o sistema imune reconhece aquele vírus. A proteína utilizada no estudo foi a Hemaglutinina (HA), pois é contra ela que os anticorpos mais eficientes são produzidos, motivo de ela ser utilizada na vacina anual.
Ao invés de um contínuo de mudanças, o mapa mostrou que os vírus tendem a se aglutinar em grupos. Mudanças na sequência muitas vezes extensas podem não representar uma diferença no reconhecimento pelos anticorpos. Ao passo que, em algumas ocasiões, mudanças pequenas na HA são suficientes para uma grande distância antigênica. Ou seja, com poucas mutações algumas Hemaglutininas podem ser bem menos reconhecidas pelos anticorpos.
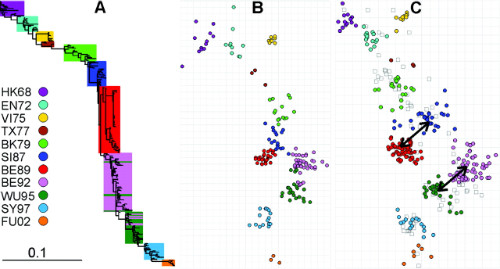
Distância genética, em A e B, e antigênica, em C, dos isolados de H3N2. Algumas linhagens embora sejam próximas geneticamente, como SI87 e BE89 em A e B, que possuem apenas um aminoácido diferente, podem não ser antigenicamente próximas, mostrado em C. Isso indica que os anticorpos produzidos contra o H3N2 em 1987 não eram eficientes contra o vírus de 1989.
Também é possível ver a mudança de resposta imune em relação ao ciclo anual. Os grupos permanecem dominantes por 3 anos em média, e o vírus que está neste grupo costuma aparecer cerca de 2 anos antes e circular por mais 2 anos depois da dominância do grupo.
Com base nestes resultados, as vacinas anuais passaram a ser pensadas de outra forma. Atualmente, a observação da resposta imune ao vírus da vacina sazonal e ao vírus circulante, além da diferença genética entre eles, é um fator importantíssimo no processo de produção. Os grupos de pesquisa que monitoram o Influenza e determinam as linhagens que farão parte da vacina precisam estar especialmente atentos a isso, pois a mudança de um grupo antigênico da Hemaglutinina de um vírus depois que ele já está sendo cultivado em ovos pode significar em uma falha na imunização da população.
Fonte:
Smith, D. (2004). Mapping the Antigenic and Genetic Evolution of Influenza Virus Science, 305 (5682), 371-376 DOI: 10.1126/science.1097211
1 Comentário » Postado em: Combate ao vírus, Resposta Imune, Vacinas


[...] o suficiente para que uma mesma pessoa seja infectada mais de uma vez, o que favorece linhagens que consigam variar o suficiente para escapar da imunidade prévia. Ao passo que, os vírus suínos encontram um hospedeiro com uma população menor, que sofre menos [...]