12/09/2009
Molécula del mes presenta: Hemaglutinina y Neuraminidasa
La Molécula del Mes es una iniciativa del banco de estructuras de proteínas RCSB PDB (A Resource for Studying Biological Macromolecules – Protein Data Bank). Mensualmente, David Goodsell escoge una estructura interesante y la describe, además de ilustrarla muy bien. Las ilustraciones están disponibles en una imagen de alta resolución que puede ser descargada.
Goodsell no sólo le sabe dar apelo estético a las imágenes, como las vuelve informativas y fáciles de entender. Acompañe estas dos proteínas del virus Influenza para ver lo que la estructura nos puede informar:
Hemaglutinina
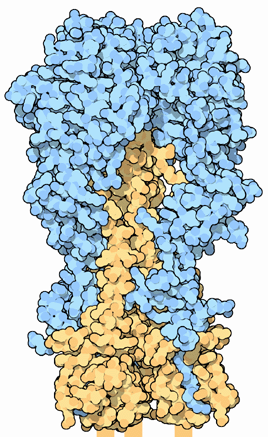
![]() La hemaglutinina (HA) es una proteína que se sitúa en la capa más externa del virus, la envoltura del vírus. Ella reconoce un azúcar de nuestra membrana celular, el ácido siálico, y es la responsable por el reconocimiento y unión del virus a nuestras células del sistema respiratorio.
La hemaglutinina (HA) es una proteína que se sitúa en la capa más externa del virus, la envoltura del vírus. Ella reconoce un azúcar de nuestra membrana celular, el ácido siálico, y es la responsable por el reconocimiento y unión del virus a nuestras células del sistema respiratorio.
Su nombre viene de esta capacidad de reconocer y unirse a las células y aglutinar hematíes (los glóbulos rojos de la sangre), una de las primeras pruebas desarrolladas para diagnosticar el virus. Su numeración es dada con base en la variación de los aminoácidos y son conocidos más de 15 tipos de HA, siendo H1, H2 y H3 las más comunes en virus que infectan a los humanos.
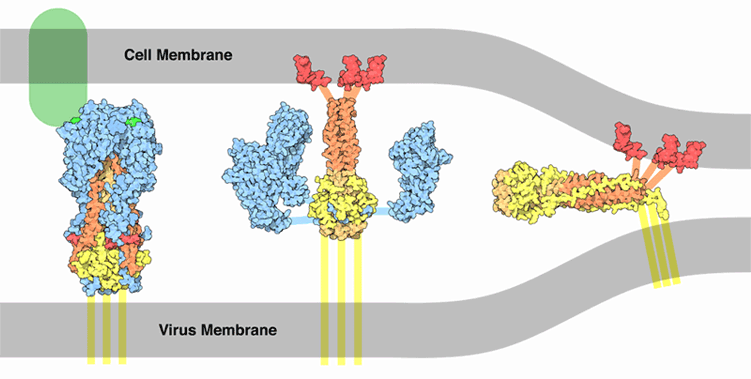
Ella reconoce nuestro ácido siálico por la porción verde mostrada en la figura de arriba, y se une a él. Con esto, el virus es endocitado por la célula, o sea, envuelto por la membrana y llevado dentro de una vesícula. La célula intenta digerir el contenido de la vesícula disminuyendo el pH dentro de la vesícula, pero esto induce un cambio en el formato de la hemaglutinina, exponiendo la región en rojo que posee mucha afinidad por la membrana de la vesícula, con esto la hemaglutinina se tuerce y acerca la membrana de la envoltura del virus, fundiendo ambas y liberando el virus de la vesícula.
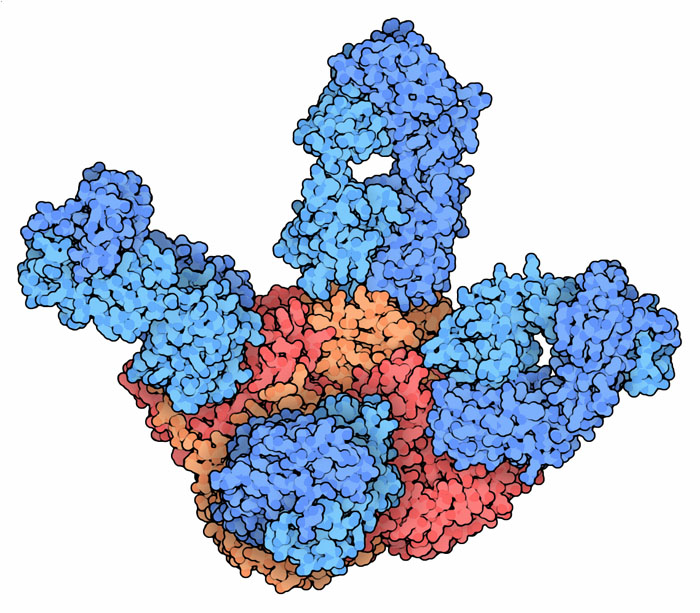
Neuraminidasa relacionada al inhibidor
Neuraminidasa
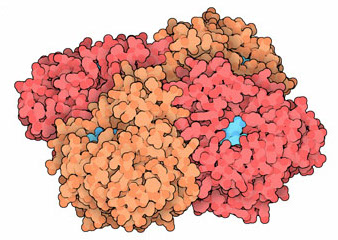
La neuraminidasa (NA) reconoce la misma molécula que la hemaglutinina, el ácido siálico de la membrana celular. Pero realiza su función de manera opuesta, su papel es ayudar al virus a dejar la célula invadida.
La neuraminidasa es necesaria para remover el ácido de la célula y permitir que el virus recién sintetizado consiga brotar para invadir la próxima célula. Por esto, ella también se ubica en la envoltura del virus, y es la segunda proteína más común, después de la hemaglutinina. También es clasificada de acuerdo con su variedad, y son conocidas 9, siendo que N1 y N2 son las más frecuentes en humanos.
Su capacidad de reconocer y cortar el ácido siálico es comprometida por inhibidores, que usamos como drogas antivirales, como la molécula en azul claro, atacando la NA del virus H5N1. Por estar en la superficie del virus, es uno de los blancos más comunes de anticuerpos, mostrados en azul en la figura de arriba. Su variación causada por mutaciones en el material genético de la influenza es el que permite que nuevas variantes dejen de ser reconocidas por el sistema inmune.
El reconocimiento de los azúcares de membrana de los diferentes tipos de huéspedes, el papel en la entrada y salida de la célula infectada, el ataque del sistema inmune y de antivirales son algunas de las características que dan la importancia de la hemaglutinina y de la neuraminidasa, que son esenciales para entender la gripe y serán visitantes frecuentes de este blog.
Fuentes:
Wiley, D., & Skehel, J. (1987). The Structure and Function of the Hemagglutinin Membrane Glycoprotein of Influenza Virus Annual Review of Biochemistry, 56 (1), 365-394 DOI: 10.1146/annurev.bi.56.070187.002053
Skehel, J., & Wiley, D. (2000). RECEPTOR BINDING AND MEMBRANE FUSION IN VIRUS ENTRY: The Influenza Hemagglutinin Annual Review of Biochemistry, 69 (1), 531-569 DOI: 10.1146/annurev.biochem.69.1.531
Colman, P., Varghese, J., & Laver, W. (1983). Structure of the catalytic and antigenic sites in influenza virus neuraminidase Nature, 303 (5912), 41-44 DOI: 10.1038/303041a0
6 Comentarios » Postado en: Uncategorized


Considero muy novedosa y actualizada la búsqueda que nos ofrecen. Quisiera saber(pués no hace mucho que leí), acerca de la posibilidad de la aparición de una mutante de HA que hiciera resistente al TAMIFLU al virus A H1N1.
Gracias por anticipado
Crescenzio,
Sí, este virus puede desarrollar resistencia y lo ha hecho, con mutaciones en la neuraminidasa. Sin embargo, el tipo de resistencia se desarrolla también lo deja más susceptible a los inhibidores de otros NA, zanamivir. Trato este tema en los textos siguientes del blog.
Saludos,
Atila
MUY INTERESANTE LA EXPLICACIPON GRACIAS POR LA PUBLICACION DE ESTE MATERIAL
[...] Los antígenos virales más importantes en relación con la producción de inmunidad protectora son la hemaglutinina externa (HA) y la glicoproteína neuraminidasa (NA). Los virus A y B de la influenza se encuentran en evolución continua produciendo nuevos tipos de [...]
[...] ya vimos antes, quien determina el tipo de célula que será infectada por el Influenza es la Hemaglutinina (HA), debido a su preferencia por el ácido siálico de la célula. Pero no es cualquier ácido [...]
[...] no se confirmó. Fue necesario adicionar al virus humanos más características de virus aviarios, Hemaglutinina (HA) y Neuraminidasa (NA), induciendo el virus a utilizar más el ácido siálico α2,3 para que él diminuyese el [...]